Analyse d'image pour l'étude de la dynamique de cellules souches
2004
Master Diploma
Project: 00085
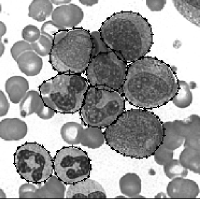
Selon des études récentes, certaines cellules souches présentes dans la moelle osseuse adulte pourraient produire des cellules différentes et offrent une perspective thérapeutique enthousiasmante. Il est donc vital de bien connaître les comportements de ces cellules souches et leurs interactions. Pour ce faire des chercheurs de ISREC étudient leur division cellulaire à travers des séquences d'images microscopiques.
En raison du grand nombre de cellules en mouvement non organisé, il est impossible pour une personne de savoir combien de divisions cellulaires ont eu lieu en un temps donné.
Le but du projet est d'étudier et de réaliser un programme d'analyse d'images qui soit capable de compter le nombre de divisions cellulaires. Le programme fera appel à une méthode de traitement d'image dite Watershed qui permettra de séparer les cellules en contact dans la première image et à une méthode de contours actifs (Snake) capable de s'accrocher automatiquement au contour des cellules par une procédure d'optimisation. Les contours actifs devront permettre de suivre les déformations et les déplacements d'une cellule à travers toute la séquence d'images.
Le programme, codé en Java, fera l'objet d'un plugin ImageJ.
Collaboration: Nathalie Garin, Institut Suisse de Recherche Expérimentale sur le Cancer (ISREC)
En raison du grand nombre de cellules en mouvement non organisé, il est impossible pour une personne de savoir combien de divisions cellulaires ont eu lieu en un temps donné.
Le but du projet est d'étudier et de réaliser un programme d'analyse d'images qui soit capable de compter le nombre de divisions cellulaires. Le programme fera appel à une méthode de traitement d'image dite Watershed qui permettra de séparer les cellules en contact dans la première image et à une méthode de contours actifs (Snake) capable de s'accrocher automatiquement au contour des cellules par une procédure d'optimisation. Les contours actifs devront permettre de suivre les déformations et les déplacements d'une cellule à travers toute la séquence d'images.
Le programme, codé en Java, fera l'objet d'un plugin ImageJ.
Collaboration: Nathalie Garin, Institut Suisse de Recherche Expérimentale sur le Cancer (ISREC)
- Supervisors
- Daniel Sage, daniel.sage@epfl.ch, 021 693 51 89, BM 4.135
- Michael Unser, michael.unser@epfl.ch, 021 693 51 75, BM 4.136