Déconvolution en microscopie dynamique
2005
Master Diploma
Project: 00115
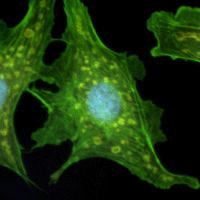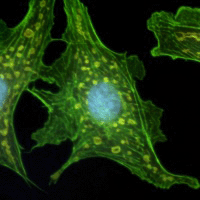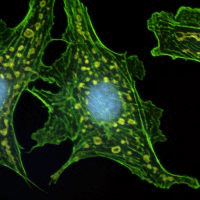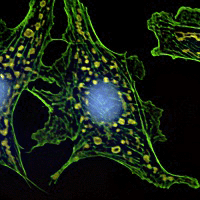
Les développements récents en biologie moléculaire nécessitent de pouvoir visualiser in vivo les déplacements des cellules ou les mouvements intracellulaires. La microscopie dynamique permet d'imager des échantillons en mouvement. Mais, comme les microscopes optiques ont une profondeur de champ limité, il est difficile de maintenir dans le plan focal les échantillons qui se déplacent en trois dimensions.
Les biologistes ont alors recourt à des algorithmes de déconvolution d'images qui tentent de restaurer les images pour fournir une image nette.
Plusieurs méthodes de déconvolution d'images ont été proposées, mais la recherche visant à améliorer les algorithmes de déconvolution est toujours active.
Dans ce projet, nous nous proposons d'étudier la déconvolution pour l'imagerie microscopique en biologie. Dans un premier temps, il s'agira d'établir un modèle paramètrable du dispositif optique donnant la fonction d'appareil (réponse impulsionnelle, PSF) à partir de lois de l'optique géométrique, puis de caractériser le bruit. Cette étude permettra de déterminer les algorithmes de déconvolution par filtrage à implémenter. L'étudiant pourra programmer plusieurs méthodes de déconvolution dont une méthode classique (Van Cittert) qui servira de référence pour établir une comparaison avec d'autres méthodes plus élaborées.
Les biologistes ont alors recourt à des algorithmes de déconvolution d'images qui tentent de restaurer les images pour fournir une image nette.
Plusieurs méthodes de déconvolution d'images ont été proposées, mais la recherche visant à améliorer les algorithmes de déconvolution est toujours active.
Dans ce projet, nous nous proposons d'étudier la déconvolution pour l'imagerie microscopique en biologie. Dans un premier temps, il s'agira d'établir un modèle paramètrable du dispositif optique donnant la fonction d'appareil (réponse impulsionnelle, PSF) à partir de lois de l'optique géométrique, puis de caractériser le bruit. Cette étude permettra de déterminer les algorithmes de déconvolution par filtrage à implémenter. L'étudiant pourra programmer plusieurs méthodes de déconvolution dont une méthode classique (Van Cittert) qui servira de référence pour établir une comparaison avec d'autres méthodes plus élaborées.
- Supervisors
- Cédric Vonesch, cedric.vonesch@epfl.ch, 021 693 51 43, BM 4.141
- Michael Unser, michael.unser@epfl.ch, 021 693 51 75, BM 4.136
- Francois Aguet