Applications des contours actifs à la trajectométrie d'un museau de souris
2006
Master Diploma
Project: 00140
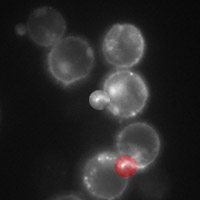
Dans le contexte de défense de l'organisme contre des cellules tumorales, on étudie le mécanisme de destruction de cellules cibles (tumorales) par des lymphocytes T spécialisées (NT-Killer). Celui-ci qui passe par une interaction entre plusieurs cellules via des protéines exprimées en leur
membrane plasmatique (pourtour de la cellule). Le contact de ces cellules implique la formation d'un "cap" sur la ou les cellules que l'on détecter. Il s'agit donc de pouvoir isoler individuellement chaque cellule, de déterminer précisément son contour en 2D voire 3D et d'estimer le pourcentage de marquage qui se concentre au point d'interaction entre cellules.
L'étudiant devra mettre en uvre des algorithmes d'analyse d'image, tels que, la transformée de Hough, des contours actifs pour déterminer finement le profil sur le contour de chaque cellule. L'implémetation sera réaliser en Java pour ImageJ.
Collaboration: Claude Bonnard, ISREC, Lausanne
- Supervisors
- Philippe Thévenaz, philippe.thevenaz@epfl.ch, 021 693 51 61, BM 4.137
- Michael Unser, michael.unser@epfl.ch, 021 693 51 75, BM 4.136