Deconvolution in 3D: an ImageJ plugin
2007
Master Diploma
Project: 00161
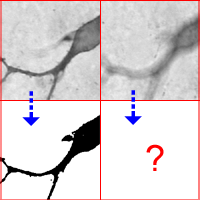
Les images de microscopie optique ont souvent une partie de l'échantillon non focalisée en raison de la profondeur de champ limitée des microscopes. C'est le cas pour les images en neuroscience où les biologistes étudient le développement neuronal. Ils acquièrent des séries d'images (z-stack) pour suivre des réseaux de dendrites en 3 dimensions mais il est difficile de détecter les dentrites qui ont toujours une partie floue.
Pourtant quand les échantillons sont simples (de couleur plus ou moins uniforme) il est imaginable de segmenter l'image défocalisée en s'inspirant des algorithmes optimaux de segmentation et en y intégrant un processus de filtrage.
Le but du projet est d'entreprendre une étude de faisabilité pour la reconstruction 3D d'objet simple à partir de série d'images défocalisées. Il s'agira d'étudier une méthodologie novatrice de segmentation d'images défocalisées, de l'implémenter et de l'expérimenter sur des données synthétiques et sur des données réelles en 2D puis en 3D. Les résultats de la segmentation pourront être utilisés pour faire une reconstruction 3D de l'objet à visualiser.
Pourtant quand les échantillons sont simples (de couleur plus ou moins uniforme) il est imaginable de segmenter l'image défocalisée en s'inspirant des algorithmes optimaux de segmentation et en y intégrant un processus de filtrage.
Le but du projet est d'entreprendre une étude de faisabilité pour la reconstruction 3D d'objet simple à partir de série d'images défocalisées. Il s'agira d'étudier une méthodologie novatrice de segmentation d'images défocalisées, de l'implémenter et de l'expérimenter sur des données synthétiques et sur des données réelles en 2D puis en 3D. Les résultats de la segmentation pourront être utilisés pour faire une reconstruction 3D de l'objet à visualiser.
- Supervisors
- Cédric Vonesch, cedric.vonesch@epfl.ch, 021 693 51 43, BM 4.141
- Michael Unser, michael.unser@epfl.ch, 021 693 51 75, BM 4.136