Localisation de chromosomes à travers une série d'images de microscopie en fluorescence
Autumn 2004
Master Semester Project
Project: 00073
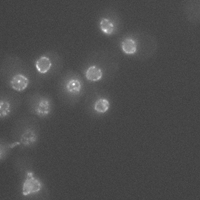
La microscopie par fluorescence permet aux biologistes d'observer l'expression de gènes et d'étudier la dynamique des chromosomes à l'intérieur d'une cellule vivante. Cette technique d'imagerie récente offre un énorme potentiel pour comprendre le fonctionnement des cellules.
L'équipe de biologistes du Prof. S. Gasser de l'Université de Genève s'intéresse en particulier à la localisation de l'extrémité d'un chromosome (télomères) à l'intérieur du noyau. Pour ce faire ils acquièrent une série d'images focales sur un microscope et ils mesurent la position des télomères par rapport aux centres du noyau.
Le but du projet est d'étudier et de développer un programme automatique d'analyse d'image pour localiser tous les noyaux et tous les télomères présents à travers la série d'images (z-stack). Le programme sera développé en Java et fera l'objet d'un plugin ImageJ qui sera remis aux biologistes de l'Université de Genève.
Collaboration: Prof S. Gasser, Département de Biologie Moléculaire, Université de Genève
L'équipe de biologistes du Prof. S. Gasser de l'Université de Genève s'intéresse en particulier à la localisation de l'extrémité d'un chromosome (télomères) à l'intérieur du noyau. Pour ce faire ils acquièrent une série d'images focales sur un microscope et ils mesurent la position des télomères par rapport aux centres du noyau.
Le but du projet est d'étudier et de développer un programme automatique d'analyse d'image pour localiser tous les noyaux et tous les télomères présents à travers la série d'images (z-stack). Le programme sera développé en Java et fera l'objet d'un plugin ImageJ qui sera remis aux biologistes de l'Université de Genève.
Collaboration: Prof S. Gasser, Département de Biologie Moléculaire, Université de Genève
- Supervisors
- Daniel Sage, daniel.sage@epfl.ch, 021 693 51 89, BM 4.135
- Michael Unser, michael.unser@epfl.ch, 021 693 51 75, BM 4.136